Alcances de un código genético sintético
Las últimas investigaciones han aprovechado la redundancia en el código genético para diseñar proteínas con aminoácidos no naturales. De manera similar, se han generado bacterias que son resistentes a la infección viral.
El ADN es reconocido por su perfección como molécula de información programable: utiliza la química de polimerización repetida para unir cuatro bloques de construcción de nucleótidos (adenina, citosina, guanina y timina) en cualquier secuencia, genera un mecanismo de copia intrínseco (apareamiento químico de bases) para moldear el reconocimiento directo y la propagación de secuencias, almacenándolas en una estructura uniforme (la doble hélice) que es fácil de leer y procesar. Las proteínas, por otro lado, son moléculas funcionales: utilizan un conjunto más grande de sustancias químicas como bloques de construcción (los 20 aminoácidos canónicos), se pliegan en estructuras distintas según la secuencia y aprovechan esa diversidad para involucrar la complejidad física del mundo tridimensional en forma de enzimas, agentes de reconocimiento y máquinas moleculares. Si bien el ADN y las proteínas tienen funciones diferentes y especializadas, la belleza (y el dogma central) de la biología molecular es que existe un vínculo directo en el que la información secuencial del primero se transcribe y se traduce en una secuencia de proteínas, residuo a residuo. Como resultado, las funciones de las proteínas se vuelven programables y evolucionables por medio de la información del ADN que las codifica y que se puede copiar y manipular fácilmente. Un estudio reciente informado por Wesley Robertson, Jason Chin y colegas lleva este concepto a un nuevo nivel.
Nuestra capacidad para programar y desarrollar secuencias de proteínas simplemente operando sobre el ADN es la razón por la que las terapias de proteínas, como los anticuerpos monoclonales, se han vuelto tan exitosas en biomedicina. A diferencia de los medicamentos de molécula pequeña, las proteínas se pueden sintetizar de manera confiable a partir de secuencias de ADN insertadas en las células, modificarse de forma predecible al cambiar el ADN que las codifica y evolucionar de manera sólida mediante la mutación del ADN y la selección de funciones terapéuticas novedosas, como la unión o modulación de una enfermedad diana. Sin embargo, a medida que crece este campo, podemos encontrarnos con una limitación fundamental en la composición y capacidad de las proteínas. A diferencia de los fármacos tradicionales, la diversidad se restringe a los 20 bloques de construcción de aminoácidos canónicos utilizados en toda la traducción proteica. Aunque la química de las proteínas se expande mediante modificaciones postraduccionales y cofactores, tales alteraciones se producen a expensas de la programabilidad, porque no se traducen directamente a partir de secuencias de ADN. Uno de los objetivos más ambiciosos de la biología sintética es rediseñar la traducción de proteínas para acomodar múltiples bloques de construcción nuevos, lo que permite la síntesis de proteínas que contienen cualquier número de estructuras químicas novedosas de nuestra elección. A medida que este esfuerzo avance, las capacidades para diseñar y generar nuevas terapias de proteínas y péptidos se irán transformando progresivamente.
¿Cómo llegamos allí? El código genético especifica la correspondencia entre una secuencia de ADN y una secuencia de proteína. En un gen codificante, cada tramo consecutivo de tres nucleótidos (un codón) corresponde a un aminoácido. Por tanto, el código genético tiene 64 codones posibles (cuatro posibilidades de nucleótidos en cada una de las tres posiciones de un codón). Entre los 64 codones, 61 son codones de sentido, cada uno de los cuales especifica 1 de los 20 aminoácidos. Los 3 codones restantes son codones de terminación, cada uno de los cuales indica la terminación de la traducción de la proteína. Entonces, en la vida tal como la conocemos, se habla por todos los codones. Pero el código genético contiene redundancia: múltiples codones a menudo especifican el mismo aminoácido. En principio, solo necesitamos 20 codones para especificar 20 aminoácidos y 1 codón de parada para la terminación de la traducción. ¿Y si pudiéramos liberar codones redundantes y reasignarlos a nuevos aminoácidos?
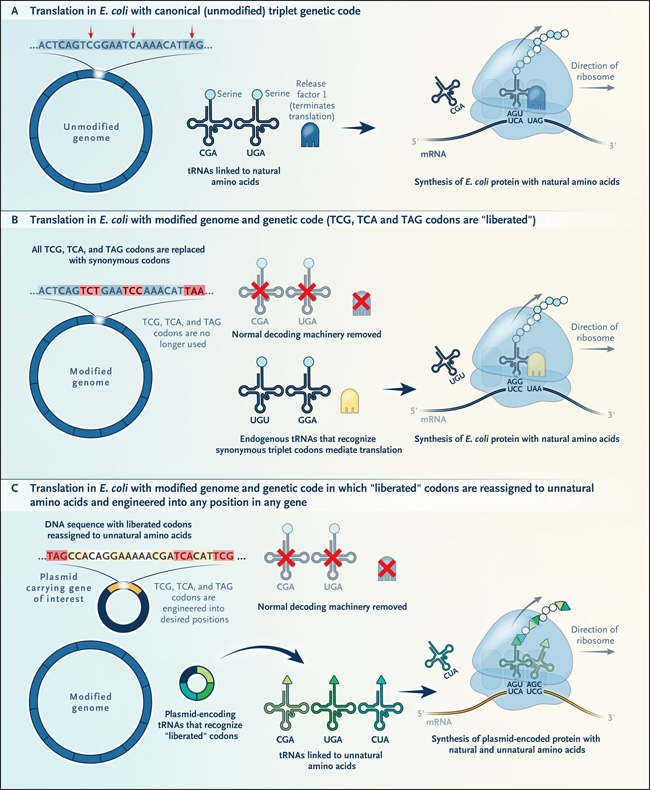
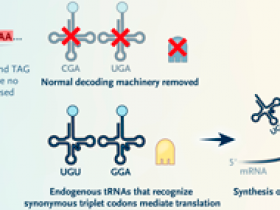
Figura 1: reescritura del código genético para crear nuevas proteínas.
La síntesis y el ensamblaje del genoma completo se utilizan para reemplazar todas las instancias de tres codones con opciones sinónimos. Normalmente, las proteínas se traducen a partir de genes que comprenden 64 codones diferentes (panel A). Previamente, en un genoma de Escherichia coli modificado, tres codones específicos (TCG, TCA y TAG) fueron reemplazados con codones sinónimos. Como tal, se puede considerar que estos tres codones han sido "liberados" y están libres para la "reasignación" (panel B). Ahora se demuestra cómo los codones "liberados" se pueden "reasignar" a aminoácidos no naturales y, por lo tanto, se pueden usar para diseñar proteínas y polímeros con químicas expandidas (panel C).
Los recientes avances técnicos en la síntesis y ensamblaje de genomas han permitido a los autores hacer exactamente eso. Previamente, habían diseñado una versión del genoma de Escherichia coli en la que reemplazaron con éxito dos codones de sentido y un codón de parada con codones sinónimos en todo el genoma (figuras 1 y figura 2). El resultado fue una cepa de E. coli carente de tres codones específicos en todo su genoma, pero que producía exactamente las mismas proteínas que la E. coli de tipo salvaje. Después de hacer evolucionar esta cepa para adaptarse mejor y eliminar los ARN de transferencia endógenos vestigiales (ARNt) y los factores que decodifican los tres codones "liberados" (figuras 1C y figura 2), asignaron cada codón liberado a un nuevo aminoácido mediante versiones diseñadas de dos moléculas: un ARNt adaptador que reconoce cada codón liberado y una enzima que une un aminoácido sintético de elección a cada ARNt. Afortunadamente, el ribosoma, la máquina molecular que une físicamente los aminoácidos de acuerdo con el orden de los codones en un gen, es capaz de tolerar los ARNt unidos a los aminoácidos no naturales. Por tanto, la nueva cepa de E. coli sintética desarrollada es totalmente capaz de sintetizar proteínas, péptidos o macrociclos no naturales que contienen múltiples aminoácidos no naturales especificados directamente por los tres codones liberados. Es muy probable que esta capacidad de acceder simultáneamente a nuevas estructuras químicas en la ingeniería y la evolución de proteínas y péptidos permita el descubrimiento de otras categorías de productos biológicos.
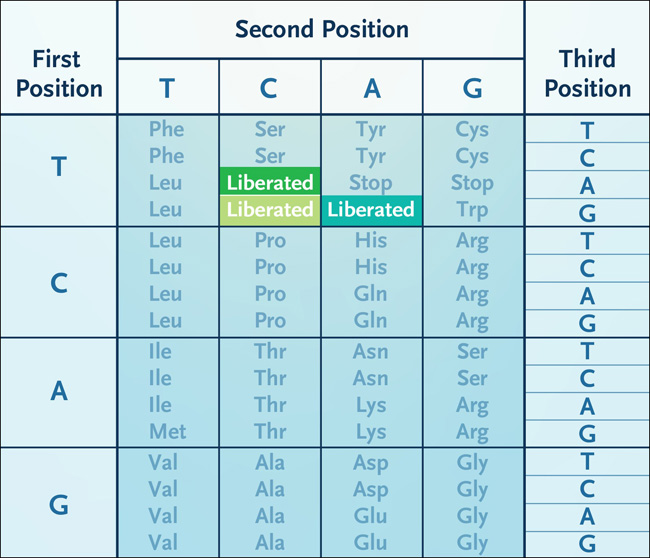
Figura 2: código genético que muestra codones liberados.
Una característica adicional de la E. coli sintética es su resistencia a la infección viral (figura 3). Si los codones liberados en ella permanecen "sin asignar" a los aminoácidos no naturales, estos se eliminan o se vuelven completamente insensatos. Cualquier gen que "use" estos codones para especificar una proteína funcional no producirá esa proteína: punto final (en el codón sin sentido). Debido a que los virus "cargan" las instrucciones para la síntesis de sus propias proteínas en los huéspedes, y esas instrucciones casi con certeza incluyen los codones ahora sin sentido, los virus se vuelven inertes. Esta característica de la resistencia viral es clínicamente valiosa porque podría proteger a la E. coli utilizada en la producción de proteínas terapéuticas de la susceptibilidad a los virus. La extensión de esta estrategia a las células de mamíferos que producen fármacos biológicos protegerá a su vez a esos organismos de la infección viral. La promesa de crear nuevos medicamentos y estrategias de resistencia viral reescribiendo el código genético fundamental es emocionante y extraordinaria.
Fuente bibliográfica
Sense and Sense Ability in a Synthetic Genetic Code
Chang C. Liu, Ph.D.
Department of Biomedical Engineering and the Center for Synthetic Biology, University of California, Irvine, Irvine.
Engl J Med 2021; 385:1045-1048